Algorytm Needlemana-Wunscha
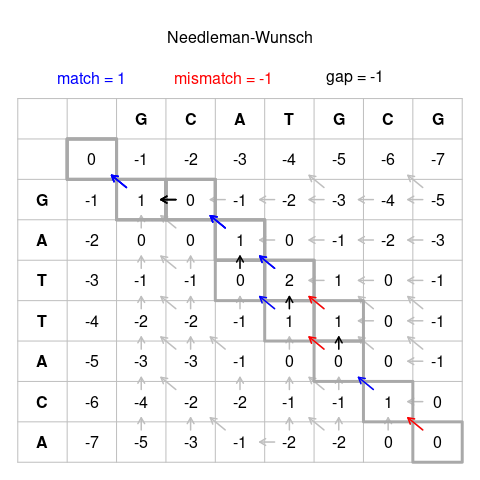
Algorytm Needlemana-Wunscha – algorytm oparty na programowaniu dynamicznym, umożliwiający znalezienie optymalnego globalnego dopasowania dwóch sekwencji.
Jest często wykorzystywany w bioinformatyce jako jedno z narzędzi do poszukiwania uliniowienia sekwencji nukleotydowych lub aminokwasowych.
Został stworzony przez Saul B. Needleman’a i Christian D. Wunsch’a oraz opublikowany w roku 1970[1]. Dzieli on większy problem obliczeniowy (np. całą sekwencję) na mniejsze problemy, i używa rozwiązań mniejszych problemów do znalezienia optymalnego rozwiązania dużego problemu[2].
Przypisy
- ↑ SAUL B.S.B. NEEDLEMAN SAUL B.S.B., CHRISTIAN D.CH.D. WUNSCH CHRISTIAN D.CH.D., A General Method Applicable to the Search for Similarities in the Amino Acid Sequence of Two Proteins, Elsevier, 1989, s. 453–463, DOI: 10.1016/b978-0-12-131200-8.50031-9, ISBN 978-0-12-131200-8 [dostęp 2020-05-27] .
- ↑ bioinformatics, [w:] Encyclopædia Britannica [dostęp 2022-09-30] (ang.).
Encyklopedie internetowe (optimization algorithm):
- Britannica: topic/Needleman-Wunsch-algorithm











