Colipasa
| Colipasa pancreática | ||||||
|---|---|---|---|---|---|---|
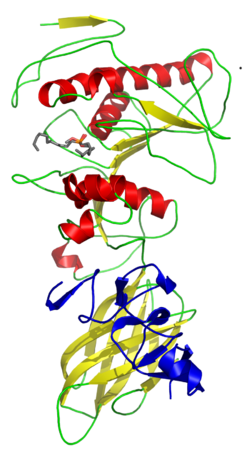 Diagrama de cintas de colipasa de cerdo (en azul) acomplejada con lipasa pancreática humana y una pequeña molécula inhibidora. | ||||||
| Estructuras disponibles | ||||||
| PDB | Buscar ortólogos: PDBe, RCSB | |||||
| Identificadores | ||||||
| Símbolo | CLPS (HGNC: 2085) | |||||
| Identificadores externos | ||||||
| Locus | Cr. 6 p21.1-ter | |||||
| ||||||
| Ortólogos | ||||||
| Especies |
| |||||
| Entrez |
| |||||
| UniProt |
| |||||
| RefSeq (ARNm) |
| |||||
| ||||||
[editar datos en Wikidata] | ||||||
La Colipasa es un cofactor proteico necesario para la actividad óptima de la lipasa pancreática. Se secreta en el páncreas exócrino en su forma inactiva, la procolipasa, y se activa en el lumen intestinal por acción de la tripsina. Su función es prevenir los efectos inhibidores de las sales biliares sobre la actividad de la lipasa pancreática, facilitando de esta forma la hidrólisis intraduodenal, de los triglicéridos de cadena larga presentes en la alimentación.
En humanos, la colipasa se encuentra codificada en el gen HGNC CLPS .[1]
Dominios proteicos
Colipasa es también una familia de proteínas evolutivamente relacionadas.
Colipasa es un pequeño cofactor proteico necesario para que la lipasa pancreática realice una hidrólisis eficiente de los triglicéridos adquiridos con los alimentos. La absorción eficiente de las grasas incluidas en la dieta es dependiente de la acción del a lipasa pancreática.
La colipasa se une al dominio C-terminal, que es el dominio no catalítico de la lipasa, estabilizándola en una conformación activa y aumentando considerablemente la hidofobicidad del sitio de unión. Numerosos estudios de la estructura del complejo y de la colipasa sola han revelado la funcionalidad de esta estructura.[2][3]
La colipasa es una proteína pequeña, con cinco enlaces disulfuro internos evolutivamente muy conservados. Se han reconocidos analogías estructurales entre diferentes proteínas: el dominio C-terminal de la lipasa pancreática; el dominio N-terminal de las lipooxigenasas, y el dominio C-terminal de la alfatoxina. Estos dominios no catalíticos son evolutivamente importantes en las enzimas más tardías para su interacción con la membrana plasmática. No se ha establecido fehacientemente si estos dominios se encuentran también implicados en una eventual unión a cofactores como es el caso de la lipasa pancreática.[3]
|
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Referencias
- ↑ Davis RC, Xia YR, Mohandas T, Schotz MC, Lusis AJ (mayo de 1991). «Assignment of the human pancreatic colipase gene to chromosome 6p21.1 to pter». Genomics 10 (1): 262-5. PMID 2045105. doi:10.1016/0888-7543(91)90509-D.
- ↑ Lowe ME (1997). «Structure and function of pancreatic lipase and colipase». Annu. Rev. Nutr. 17: 141-158. PMID 9240923. doi:10.1146/annurev.nutr.17.1.141.
- ↑ a b Verger R, van Tilbeurgh H, Cambillau C, Bezzine S, Carriere F (1999). «Colipase: structure and interaction with pancreatic lipase». Biochim. Biophys. Acta 1441 (2–3): 173-184. PMID 10570245.
- ↑ Egloff MP, Marguet F, Buono G, Verger R, Cambillau C, van Tilbeurgh H (marzo de 1995). «The 2.46 A resolution structure of the pancreatic lipase-colipase complex inhibited by a C11 alkyl phosphonate». Biochemistry 34 (9): 2751-62. PMID 7893686. doi:10.1021/bi00009a003.
Lecturas adicionales
- Weyrich P, Albet S, Lammers R, et al. (2009). «Genetic variability of procolipase associates with altered insulin secretion in non-diabetic Caucasians». Exp. Clin. Endocrinol. Diabetes 117 (2): 83-7. PMID 18726866. doi:10.1055/s-2008-1078733.
- Crandall WV, Lowe ME (2001). «Colipase residues Glu64 and Arg65 are essential for normal lipase-mediated fat digestion in the presence of bile salt micelles». J. Biol. Chem. 276 (16): 12505-12. PMID 11278590. doi:10.1074/jbc.M009986200.
- Miled N, Canaan S, Dupuis L, et al. (2000). «Digestive lipases: from three-dimensional structure to physiology». Biochimie 82 (11): 973-86. PMID 11099794. doi:10.1016/S0300-9084(00)01179-2.
- van Tilbeurgh H, Egloff MP, Martinez C, et al. (1993). «Interfacial activation of the lipase-procolipase complex by mixed micelles revealed by X-ray crystallography». Nature 362 (6423): 814-20. PMID 8479519. doi:10.1038/362814a0.
- Wermter AK, Scherag A, Holter K, et al. (2009). «Procolipase gene: no association with early-onset obesity or fat intake». Obes Facts 2 (1): 40-4. PMID 20054203. doi:10.1159/000196379.
- Lindner I, Helwig U, Rubin D, et al. (2005). «Putative association between a new polymorphism in exon 3 (Arg109Cys) of the pancreatic colipase gene and type 2 diabetes mellitus in two independent Caucasian study populations». Mol Nutr Food Res 49 (10): 972-6. PMID 16189801. doi:10.1002/mnfr.200500087.
- Sims HF, Lowe ME (1992). «The human colipase gene: isolation, chromosomal location, and tissue-specific expression». Biochemistry 31 (31): 7120-5. PMID 1643046. doi:10.1021/bi00146a013.
- Lowe ME, Rosenblum JL, McEwen P, Strauss AW (1990). «Cloning and characterization of the human colipase cDNA». Biochemistry 29 (3): 823-8. PMID 2337598. doi:10.1021/bi00455a032.
- van Tilbeurgh H, Bezzine S, Cambillau C, et al. (1999). «Colipase: structure and interaction with pancreatic lipase». Biochim. Biophys. Acta 1441 (2–3): 173-84. PMID 10570245.
- D'Silva S, Xiao X, Lowe ME (2007). «A polymorphism in the gene encoding procolipase produces a colipase, Arg92Cys, with decreased function against long-chain triglycerides». J. Lipid Res. 48 (11): 2478-84. PMID 17715423. doi:10.1194/jlr.M700371-JLR200.
- Gerhard DS, Wagner L, Feingold EA, et al. (2004). «The Status, Quality, and Expansion of the NIH Full-Length cDNA Project: The Mammalian Gene Collection (MGC)». Genome Res. 14 (10B): 2121-7. PMC 528928. PMID 15489334. doi:10.1101/gr.2596504.
- Sternby B, Engström A, Hellman U, et al. (1984). «The primary sequence of human pancreatic colipase». Biochim. Biophys. Acta 784 (1): 75-80. PMID 6691986. doi:10.1016/0167-4838(84)90175-4.
- Sias B, Ferrato F, Grandval P, et al. (2004). «Human pancreatic lipase-related protein 2 is a galactolipase». Biochemistry 43 (31): 10138-48. PMID 15287741. doi:10.1021/bi049818d.
- Lowe ME (1997). «Structure and function of pancreatic lipase and colipase». Annu. Rev. Nutr. 17: 141-58. PMID 9240923. doi:10.1146/annurev.nutr.17.1.141.
- Strausberg RL, Feingold EA, Grouse LH, et al. (2002). «Generation and initial analysis of more than 15,000 full-length human and mouse cDNA sequences». Proc. Natl. Acad. Sci. U.S.A. 99 (26): 16899-903. PMC 139241. PMID 12477932. doi:10.1073/pnas.242603899.
- Sugar IP, Mizuno NK, Momsen MM, et al. (2003). «Regulation of lipases by lipid-lipid interactions: implications for lipid-mediated signaling in cells». Chem. Phys. Lipids 122 (1–2): 53-64. PMID 12598038. doi:10.1016/S0009-3084(02)00178-0.
- van Tilbeurgh H, Sarda L, Verger R, Cambillau C (1992). «Structure of the pancreatic lipase-procolipase complex». Nature 359 (6391): 159-62. PMID 1522902. doi:10.1038/359159a0.
- Davis RC, Xia YR, Mohandas T, et al. (1991). «Assignment of the human pancreatic colipase gene to chromosome 6p21.1 to pter». Genomics 10 (1): 262-5. PMID 2045105. doi:10.1016/0888-7543(91)90509-D.
Enlaces externos
- MeSH: Colipases (en inglés)
 Datos: Q3682656
Datos: Q3682656










